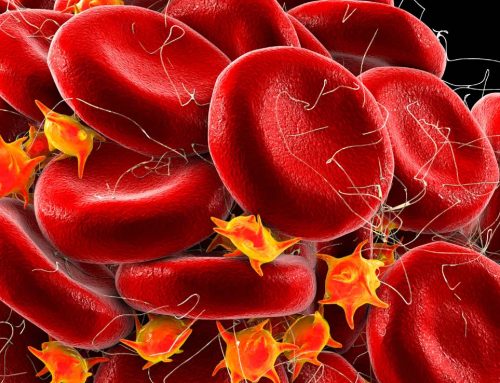
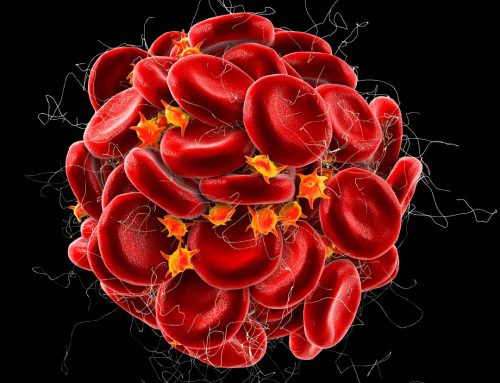
Células cardíacas derivadas de células iPS são mostradas. Um único par de células de DNA do gene PRKAG2 foi editado, utilizando o método desenvolvido pelos doutores Miyaoka e Conklin.
O estudo publicado na revista Nature Methods, descreve um meio preciso e eficiente de capturar mutações genéticas raras que causam doenças, bem como a forma de corrigi-las .
“Os avanços na genética humana levaram à descoberta de centenas de alterações genéticas ligadas a doenças, mas até agora não tínhamos um meio eficiente de estudá-las”, explicou o doutor Bruce Conklin. “Temos que ter a capacidade de projetar o genoma humano, letra após letra, com ferramentas que sejam eficientes, robustas e precisas. O método que descrevemos em nosso estudo faz exatamente isso.”
Um dos principais desafios que impedem os investigadores de estudar certas doenças genéticas de forma eficiente é que elas podem ocorrer raramente, cerca de 1%, tornando difícil a tarefa de encontrá-las e estudá-las.
“Para o nosso método de trabalho, precisávamos encontrar uma maneira de identificar uma única mutação entre centenas de células normais, saudáveis”, acrescentou o cientista Yuichiro Miyaoka , Ph.D., autor do artigo. “Então, nós desenvolvemos uma sonda fluorescente especial que pode distinguir a sequência mutante das sequências originais. Assim, fomos capazes de separar as células modificadas nos dois grupos de sequências, mesmo quando elas são muito pequenas, uma em cada mil células. Este é um nível de sensibilidade mais de cem vezes maior do que os métodos tradicionais”.
“A engenharia genômica confronta-se com um desafio logístico: alta fidelidade e precisão de resultados de mutagênese em eventos mutagênicos raros. Mas isolar uma rara mutação celular sem resistência a antibióticos é muito mais difícil”, escreveram os pesquisadores em seu artigo (“Isolation of single-base genome-edited human iPScells without antibiotic selection”). “Para ajudar a resolver este problema, foi desenvolvido um método que permite a detecção eficiente de uma mutação, o sib-selection (originalmente um método de clonagem de levedura ) e o isolamento de raros clones com a mutação desejada . Foi utilizado o PCR digital droplet recentemente desenvolvido (ddPCR) e as condições de crescimento de células iPS adaptadas para que os clones mutantes raros pudessem ser isolados com eficiência sem precedentes. Nós usamos o nosso método para introduzir mutações pontuais associadas a doenças em cinco genes (PHOX2B, pKP2 , RBM20, PRKAG2 e BAG3) . Para cada gene, nós projetamos um par de TALE nuclease (TALENs) ou um guia de RNA (gRNA) para direcionar as sequências próximas ao local de mutação desejado e um 60-nucleotide (nt) de filamento único (single-stranded) doador de DNA, contendo a mutação ” .
“Nosso método fornece uma nova maneira de captar e amplificar mutações específicas que normalmente são extremamente raras”, continuou o Dr. Conklin . “Este método de alta eficiência e alta fidelidade poderia muito bem ser a base para a próxima fase da pesquisa genética humana.”
Fonte: Genetic Engineering & Biotechnology News